Una lucha con base
Científicos de Exactas UBA crearon TuberQ, una base de datos online, de acceso libre y gratuito, para mejorar la selección de los blancos en el desarrollo de drogas contra la tuberculosis. La enfermedad ataca, cada año, a nueve millones de personas en todo el planeta, según la Organización Mundial de la Salud.

“La base de datos ayuda a los científicos a avanzar en su investigación en forma más rápida al tener la información ordenada y con pistas para no meterse en callejones sin salida cuando se piensa en el diseño de drogas contra la tuberculosis”, precisa Adrián Turjanski, director del proyecto TuberQ.
http://noticias.exactas.uba.ar/audio/AdrianTurjanski.mp3
Descargar archivo MP3 de Adrián Turjanski
Todo lo que conviene saber a la hora de diseñar medicamentos para combatir mejor la tuberculosis, que enferma cada año a nueve millones de personas según la Organización Mundial de la Salud (OMS), es posible hallarlo en TuberQ. Se trata de una base de datos gratuita, de reciente acceso por Internet, elaborada por científicos argentinos de la Facultad de Ciencias Exactas y Naturales de la Universidad de Buenos Aires (Exactas UBA) tras dos años de intenso trabajo.
Con sólo clickear en http://tuberq.proteinq.com.ar es posible contar con una puesta al día de todo lo que hoy se conoce sobre la bacteria con forma de bacilo que descubrió Robert Koch. Y también acceder a un análisis basado en algoritmos bioinformáticos que muestra los caminos más adecuados a seguir para dar con mejores blancos proteicos hacia los cuales apuntar para el desarrollo de drogas contra esta infección respiratoria.
“La base de datos ayuda a los científicos a avanzar en su investigación en forma más rápida al tener la información ordenada y con pistas para no meterse en callejones sin salida cuando se piensa en el diseño de drogas contra la tuberculosis”, precisa Adrián Turjanski, director del proyecto TuberQ, desde su laboratorio en Exactas.
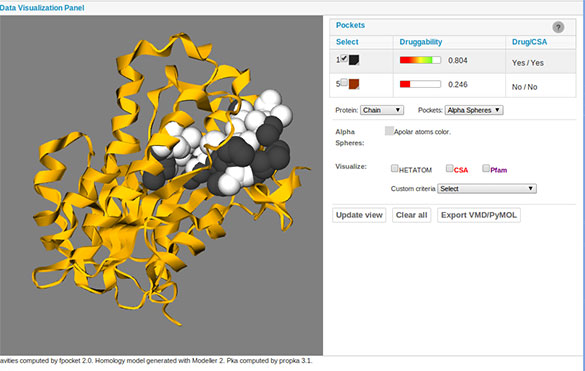
No es la única base de este tipo en el mundo, pero posee características que la distinguen del resto. “TuberQ tiene la peculiaridad de contar con un análisis de la estructura de las proteínas de la bacteria y de sus propiedades. Por ejemplo, hay una proteína que originalmente parecía buena para trabajar pero, cuando la estudiamos, vimos que no tenía agujero para unir drogas, es decir no era factible”, describe este especialista en simular, en programas de computación, cómo funcionan distintas proteínas.
En su trabajo cotidiano de alquimista cibernético, Turjanski -que dirige el Grupo de Bioinformática Estructural en el Instituto de Química Física de los Materiales, Medio Ambiente y Energía (INQUIMAE/ CONICET)- somete virtualmente a las proteínas a diversas situaciones en sus programas de computación y ensaya respuestas posibles. “Nos hacemos preguntas sobre el funcionamiento de las proteínas, sobre cómo inhibirlas, qué mutaciones las afectan, y eso lo vinculamos con el desarrollo de fármacos para enfermedades”, grafica. Los resultados de estas simulaciones luego son analizados en el terreno experimental.
A un click
Con sólo ingresar al banco de datos, que cuenta con un tutorial guía para quienes lo visitan por primera vez, se abre todo el abanico de datos que conviene saber sobre la tuberculosis a nivel de proteínas: cuáles son las drogas que hoy se usan, qué se experimentó, qué camino conviene transitar y cuál obviar. Cuenta, además, con imágenes de proteínas que permiten ver cuáles serían los mejores sitios por donde ingresar con medicación para debilitar -hasta exterminar- a la bacteria que produce la tuberculosis.
“Nuestro objetivo es comprender o elegir mejor cómo vamos a atacar a la bacteria de la tuberculosis”, precisa Turjanski, profesor del Departamento de Química Biológica de Exactas. Y más adelante detalla parte del proceso que habitualmente tiene lugar en el desarrollo de drogas de diseño, en el “que interviene cada vez más el uso de experimentos computacionales. Parece de ciencia ficción pero, en parte, es lo que estamos haciendo”, observa.
¿De qué se trata este proceso? “Tomamos una muestra de una bacteria, que se pasa a un secuenciador para obtener el genoma completo –indica-. Toda la información genética se convierte en letras dentro un algoritmo bioinformático, que en la computadora se transforman en genes. Cada uno de sus genes es dividido en sus dominios y su estructura es modelada. En consecuencia, por un lado, se tiene un montón de genes y estructuras. Por otro lado, todos los genes y funciones son mapeadas a las vías metabólicas del bacilo. Entonces, uno sabe cómo interactúa y qué rol cumple cada uno dentro de la célula. Por suerte, como hay numerosos experimentos hechos en tuberculosis, cada uno de esos genes fue reportado en algún experimento de alguna manera. Nosotros vinculamos toda esa información”.
Todo este material está disponible hoy y será actualizado en forma permanente en la base de datos. “Un paso más que también hacemos, es lo que llamamos librería de drogas de posibles compuestos líderes”, señala. Esto permite simular cómo actuarían ciertas drogas en determinados blancos, y luego todo debe ser probado en experimentos de laboratorio. “No siempre hay éxito –aclara-, a veces la realidad sorprende para bien o para mal”.
A pesar de que TuberQ recientemente fue publicada en Internet, ya ha tenido repercusiones. “Se ha acercado gente a la que le gustó el análisis que hemos realizado y nos pidieron que trabajáramos en una base de datos para otros patógenos”, concluye.