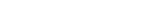La fagoseñal
Un equipo de investigación desarrolló un método sencillo, rápido y de bajo costo para el diagnóstico de la tuberculosis, usando bacteriófagos que permiten detectar en las muestras de pacientes la presencia de la micobacteria que produce la enfermedad, además de monitorear la eficacia de los tratamientos e identificar cepas resistentes a antibióticos. La bióloga Mariana Piuri, que lidera el proyecto, acaba de ser galardonada con el premio Milstein.
La tuberculosis, una antigua enfermedad con la que la humanidad convive hace miles de años, sigue siendo uno de sus principales dilemas epidemiológicos. Se estima que cada día fallecen en todo el mundo unas 3.500 personas por esta patología infecciosa que se transmite a través del aire, cuando un infectado tose o estornuda. Y como ocurre con casi todas las llamadas “enfermedades de la pobreza”, cuya prevalencia está directamente ligada a condiciones de vida precarias, un desafío central que plantea la tuberculosis es la ausencia de mecanismos más eficaces para detectarla: apenas en el 36% de los casos se arriba a un diagnóstico efectivo.
En este marco, un equipo de investigación de Exactas UBA liderado por la bióloga Mariana Piuri desarrolló un método sencillo, rápido y de bajo costo para el diagnóstico de la tuberculosis, que no sólo revela la presencia de la bacteria que produce la infección, sino que además permite monitorear la eficacia del tratamiento administrado a cada paciente, una herramienta clave para identificar fallas terapéuticas, prevenir recaídas y disminuir el riesgo de desarrollar una enfermedad resistente a los antibióticos. Por este avance, Piuri acaba de ser galardonada con el premio Milstein.
“La base de este desarrollo es el trabajo que venimos haciendo desde hace años con bacteriófagos, que son virus que infectan a las bacterias. Lo que hicimos fue modificar genéticamente estos bacteriófagos, introduciéndoles un gen que codifica un reportero fluorescente, de manera tal que cuando se encuentran con la micobacteria que produce la tuberculosis, la infectan, y la bacteria produce una proteína fluorescente. Entonces uno puede verla fácilmente en la muestra de esputo de un paciente utilizando un microscopio”, explica Piuri, directora del Laboratorio de Bacteriófagos y Aplicaciones Biotecnológicas que funciona en el Instituto de Química Biológica de la Facultad de Ciencias Exactas y Naturales (IQUIBICEN, UBA – CONICET).
Si la bacteria se ve en el microscopio de un color rojo fluorescente, eso significa que en el esputo hay Mycobacterium tuberculosis, el agente que produce la infección.
El kit se llama FluoTB y se presenta como una alternativa innovadora, rápida y accesible para diagnosticar la enfermedad sin necesidad de realizar cultivos. Y reúne diversas ventajas respecto de otros métodos diagnósticos, como los tests genotípicos y la baciloscopía.
En comparación con las técnicas tradicionales, el procedimiento es sencillo. Se toma una muestra del esputo del paciente, se la somete a un breve proceso para eliminar otros microorganismos que puedan estar presentes en la muestra, y se la pone en contacto con los bacteriófagos modificados. Si la bacteria se ve en el microscopio de un color rojo fluorescente, eso significa que en el esputo hay Mycobacterium tuberculosis, el agente que produce la infección. “Y el resultado –señala Piuri– puede estar disponible de tres a cinco días desde la toma de la muestra”.
Un dato central es que los fagos sólo infectan bacterias “viables”, puntualiza la investigadora del CONICET. Los métodos genotípicos detectan la presencia de ADN bacteriano pero no si las bacterias en la muestra están vivas o muertas. “La ventaja fundamental de FluoTB respecto de esos tests es precisamente la posibilidad de determinar la viabilidad de las micobacterias”.
“Y como el fago sólo infecta a la bacteria si ésta es viable –agrega Piuri–, nosotros podemos combinar el ensayo con distintas drogas que se usan para el tratamiento de la enfermedad y, dependiendo de si hay fluorescencia o no, saber si esa bacteria es sensible o resistente a distintos antibióticos. Si es resistente, seguirá viva aunque esté en contacto con la droga, y el virus va a infectarla, encendiendo la señal fluorescente”.
Entonces, además de confirmar el diagnóstico con mayor precisión, el test puede indicar si el tratamiento está funcionando e incluso revelar si las bacterias son resistentes al tratamiento. “Algunos métodos genotípicos pueden usarse para determinar la resistencia a antibióticos, pero lo que hacen es buscar las mutaciones que son responsables de otorgarle esa resistencia a la bacteria, es decir, una secuencia particular, que no puede aplicarse a todos los antibióticos que existen”.
La investigadora detalla los problemas que plantea el seguimiento de los tratamientos, y que el nuevo kit resolvería: “Cuando el paciente llega con síntomas por primera vez al hospital, se le toma una muestra y se diagnostica si tiene o no tuberculosis. Si la baciloscopía dio positiva, como las metodologías convencionales para determinar con qué antibiótico debe tratarse pueden demorar unas ocho semanas, al paciente se le administra, hasta tener el resultado de ese cultivo, un tratamiento empírico. Pensemos que el tratamiento para tuberculosis es muy largo: un mínimo de seis meses y hasta dos años de antibióticos. Cuando el paciente vuelve al laboratorio a los dos meses, hay que tomar una nueva muestra para determinar si hubo una disminución de las bacterias viables en el esputo. Para saber esto por métodos de cultivo, es necesario esperar entre cuatro y seis semanas más. El método genotípico no sirve para esto, porque el paciente podría estar respondiendo al tratamiento y puede que haya ADN bacteriano en la muestra de esputo. Es ahí donde necesitás un método que diga si la bacteria está viva o no, si es resistente a ese antibiótico y hay que cambiarlo”. FluoTB puede hacerlo en solo tres a cinco días.
La metodología ya pudo ser evaluada en unas 300 muestras de personas con tuberculosis, con muy buenos resultados.
Y esta información es fundamental: si un paciente no recibe el antibiótico adecuado, sigue contagiando y diseminando cepas resistentes. Por otra parte, estas inconsistencias terapéuticas, prolongadas en el tiempo, provocan que muchos pacientes abandonen el tratamiento, agravando el problema epidemiológico.
La estrategia ideada por Piuri también presenta ventajas respecto de la baciloscopía, el método más extendido de diagnóstico de tuberculosis –que consiste en teñir la muestra con una sustancia especial para observar la bacteria en el microscopio–, una técnica de muy baja sensibilidad que no logra detectar la enfermedad en pacientes con baja carga bacteriana. Y que tampoco discrimina entre bacterias viables y no viables.
Luego de años de investigación, el equipo del IQUIBICEN logró llevar el desarrollo a un entorno real, evaluándolo en muestras clínicas de pacientes concretos. En cooperación con el Hospital Muñiz y con el Instituto de Tisioneumonología “Prof. Dr. Raúl Vaccarezza”, la metodología ya pudo ser evaluada en unas 300 muestras de personas con tuberculosis, con muy buenos resultados y altos valores de sensibilidad y especificidad en la detección de la enfermedad.
La sencillez del test permitiría implementarlo en centros de salud con equipamiento básico, garantizando un mayor acceso de la población a este tipo de diagnósticos, en regiones donde la incidencia de la enfermedad es mayor. “La infraestructura hospitalaria que demanda es mínima –sostiene Piuri–. Por lo pronto, un microscopio de fluorescencia, que existe en muchos laboratorios que hacen diagnóstico de tuberculosis, adaptado para usarlo en el test. Y hablamos de hospitales públicos, donde se hace la gran mayoría de los diagnósticos de tuberculosis. En centros de salud privados pueden hacer una baciloscopía, pero después derivan a centros especializados”.
El grupo ya cuenta con el kit completo con todos los componentes, el formato y el escalado de la producción para llevar el desarrollo a la fase comercial.
El mes pasado, la bióloga fue distinguida por este proyecto con la 2º Mención del Premio César Milstein 2025 “a la investigación en biotecnología con impacto en la salud”, que entrega la Fundación Pablo Cassará a través del Instituto Milstein. Un fuerte espaldarazo para coronar años de esfuerzo en la forma de una herramienta concreta para la clínica médica, que llega, dice Piuri, “en un momento muy difícil. Este desarrollo venía financiándose con el respaldo de la Agencia I+D+i, hoy paralizada. De hecho, el reconocimiento del Premio Milstein no sólo nos permite visibilizar nuestro trabajo, sino también sumar algo de fondos para el laboratorio”.
Ella y otros dos miembros de su grupo de investigación –la bióloga Florencia Payaslián y el infectólogo Agustín Seijo– conformaron una startup de base científica, Herellex, que se encuentra consolidando la etapa de transferencia tecnológica y avanza –en asociación con una importante empresa nacional especializada en la fabricación de kits diagnósticos de enfermedades infecciosas– hacia la aprobación del test con vistas a comercializarlo, en el país y en la región.
A fines de agosto, el proyecto recibió un nuevo impulso cuando Herellex fue elegida ganadora de la fase provincial –por la Ciudad de Buenos Aires– en la categoría “despegue emprendedor” del concurso Emprendimiento Argentino, organizado por la Secretaría PyME, Emprendedores y Economía del Conocimiento.
“Ya tenemos el kit completo –cuenta Piuri–, con todos los componentes, el formato y el escalado de la producción para llevar el desarrollo a la fase comercial. Queda una última etapa de inversión para ya proceder al registro en la ANMAT, lo que supone una validación final, más allá de las que ya hicimos en estudios previos”.
La estadística epidemiológica indica que en la Argentina, donde el año pasado se notificaron unos 16.600 casos de tuberculosis, deberían realizarse unas 200 mil pruebas anuales para sostener un programa sólido de detección y prevención de la enfermedad. El análisis genotípico de una sola muestra cuesta hoy alrededor de 100 dólares. El nuevo kit –que aún no tiene un precio de mercado– tendría un costo mucho más accesible.
Mariana Piuri lleva una década y media en el desarrollo de esta tecnología. “Sí, es una línea de trabajo que empecé hace muchos años, cuando hacía mi posdoctorado en el Pittsburgh Bacteriophage Institute, en la Universidad de Pittsburgh”, señala. En 2010 regresó a la Argentina con el objetivo de potenciar su trabajo con un grupo de investigación local. Así nació el Laboratorio de Bacteriófagos, una apuesta por la ciencia básica que hoy empieza a dar sus frutos.