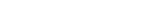
Desde el genoma hasta la droga
Mediante técnicas bioinformáticas los investigadores del equipo a cargo de Marcelo Martí intentan encontrar un compuesto líder para el desarrollo de fármacos que logren combatir la tuberculosis en su fase de latencia.
La tuberculosis es causada por Mycobacterium tuberculosis (Mt), una bacteria que posee unos 5000 genes y cuyo genoma ya ha sido secuenciado. Más conocido como bacilo de Koch, este microorganismo ingresa en el cuerpo y casi siempre afecta a los pulmones provocando la enfermedad.
Según la Organización Mundial de la Salud (OMS), una tercera parte de la población mundial tiene tuberculosis latente, es decir, son personas que están infectadas por el bacilo pero no presentan síntomas (no son enfermos) ni pueden transmitir la infección.
La forma activa de la enfermedad se puede tratar con una combinación de cuatro antibióticos administrados durante seis meses. Sin embargo, no sólo el tratamiento es demasiado largo y costoso, sino que suelen aparecer cepas que no responden a la terapia convencional y que sólo pueden tratarse con fármacos que tienen efectos secundarios graves o, incluso, otras cepas para las que no existe tratamiento. Por otra parte, no existen medicamentos que ataquen la enfermedad en la fase de latencia. Por eso, encontrar nuevos fármacos que puedan atacar el bacilo durante este período es fundamental para lograr erradicar esta enfermedad. Este es, justamente, el objetivo del proyecto que dirige el biólogo Marcelo Martí, junto a un equipo multidisciplinario.
“Nos proponemos encontrar, utilizando un enfoque dialéctico teórico-experimental, un compuesto líder para el desarrollo de fármacos antituberculínicos”, explica Martí. “Específicamente, nuestro foco está en la búsqueda de compuestos capaces de atacar al Mycobacterium Tuberculosis primordialmente en la fase de latencia. Para eso, es crucial comprender la importancia, sensibilidad y drogabilidad del proteoma del bacilo en condiciones de estrés, caracterizadas por la presencia de especies reactivas de nitrógeno y oxígeno. Nosotros utilizamos técnicas bioinformáticas que nos permiten encontrar las proteínas que son potenciales blancos moleculares sensibles a la presencia de especies reactivas de nitrógeno y oxígeno, esenciales y drogables (es decir, que posean la capacidad de ser inhibidos por compuestos tipo droga)”, explica el investigador. Los blancos con las mejores perspectivas serán luego sujetos a un protocolo de búsqueda virtual de potenciales inhibidores. Los compuestos seleccionados como potencialmente activos serán puestos a prueba en ensayos in-vitro para medir su potencia de inhibición, y en ensayos basados en célula entera (whole cell based screening assays) para medir su actividad bactericida.
Marcelo Martí es experto en bioinformática y utiliza herramientas que permiten procesar, almacenar y analizar una gran cantidad de información biológica y combinarla con estudios experimentales. Las técnicas de computación que utilizan los investigadores tienen en cuenta parámetros de la biología molecular y de la química. “El desarrollo de estas tecnologías nos permite obtener un conocimiento molecular de gran utilidad tanto para el futuro tratamiento de la tuberculosis como de otras enfermedades infecciosas”, sostiene Martí.
El enfoque propuesto busca, además, cubrir todo el rango de aplicaciones bioinformáticas, porque “va desde el genoma hasta la droga. Esto, sumado a la validación experimental in-vitro y en células enteras, representa un salto cualitativo respecto de proyectos tradicionales en esta área que, o se centran en el análisis genómico y terminan en la selección de posibles blancos, o realizan una búsqueda virtual en un blanco elegido desde la literatura, o realizan búsquedas masivas utilizando ensayos experimentales”, afirma el investigador.
Para llevar adelante la investigación, el grupo ha realizado un convenio con empresas de software (FluxIT) y de computación de alta performance (BifeSA) para los desarrollos de software bioinformático relacionado con el proyecto y en un marco más general del desarrollo bioinformático nacional.
El grupo de investigación de Martí combina y coordina la experiencia y el trabajo de grupos nacionales e internacionales que se encuentran estudiando distintos aspectos de la bioquímica y biología del bacilo de la tuberculosis y con quienes el grupo de trabajo mantiene activas colaboraciones. Está integrado, además, por investigadores con formación diversa tales como biólogos, químicos, físicos e informáticos, lo que lo hace, en sentido literal, un grupo interdisciplinario en el área de las biociencias.
La tuberculosis mata a casi dos millones de personas por año en todo el mundo. Muchas de ellas en los países en desarrollo. En Argentina, más de 12.000 personas enferman anualmente y más de 1.000 mueren a causa de esta enfermedad. El 20% de los infectados que se encuentran en fase latente desarrollarán la enfermedad. En este contexto, no caben dudas de que lograr el desarrollo de nuevos fármacos es apremiante.

Grupo de Biofisicoquímica y
Bioinformática Estructural de Proteínas
(Departamento de Química Biológica e INQUIMAE)
Laboratorios: QB65, 4to. piso y E1 (entrepiso), Pabellón II. Tel.: 4576-3378/80.
Dirección: Marcelo Adrián Martí
Integrantes: Luciana Capece
Posdoctorados: Gabriel Jara
Tesistas de doctorado: Diego Gauto, Lucas Defelipe, Victoria Dumas, Leandro Radusky, Lucía Álvarez, Claudia Ramírez, Esteban Lanzarotti
Tesistas de grado: Juan Blanco, Leandro Di-Giano, Jordana Galizia